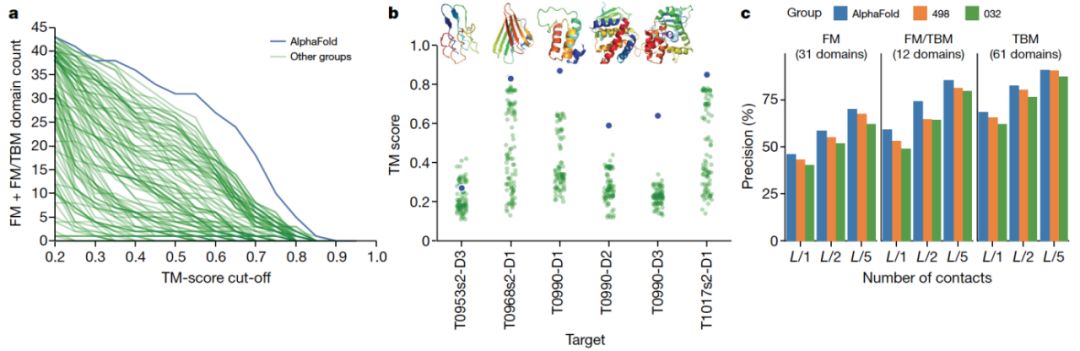
蛋白质结构预测可用于从氨基酸序列中确定蛋白质的三维形状。由于蛋白质的结构在很大程度上决定了其功能,因此这个问题具有根本的重要性;然而,蛋白质的结构很难通过实验来确定。最近通过利用遗传信息取得了相当大的进展。通过分析同源序列中的协变量是可以推断哪些氨基酸残基是接触的,这有助于蛋白质结构的预测。而与接触预测相比,英国DeepMind公司的Andrew W. Senior等人证明可以训练一个神经网络来精确预测残基对之间的距离,这传递更多关于结构的信息。利用这些信息,研究人员构造了一个能准确描述蛋白质形状的平均力势。且通过简单的梯度下降算法,可以在不需要复杂采样过程的情况下,优化得到的势能。所得的系统命名为AlphaFold,即使对于同源序列较少的序列,也能达到很高的精度。AlphaFold代表了蛋白质结构预测的一个重大进展。希望这种提高的准确性能够深入了解蛋白质的功能和功能异常,特别是在尚未通过实验确定同源蛋白质结构的情况下。
Senior, A.W., Evans, R., Jumper, J. et al. Improved protein structure prediction using potentials from deep learning. Nature (2020)
doi:10.1038/s41586-019-1923-7
https://doi.org/10.1038/s41586-019-1923-7


















