为了更好地了解在原子水平上感染的初始步骤,清华大学王新泉和张林琦等研究人员,揭示了新冠病毒(SARS-CoV-2)突刺受体结合域与ACE2受体结合的结构。
本文要点
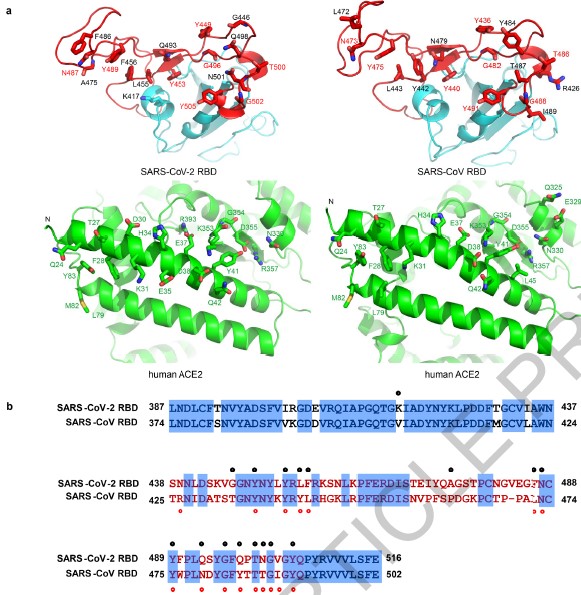
1)研究人员报道了2.45Å分辨率下与细胞受体ACE2结合的SARS-CoV-2突刺受体结合域(RBD)的晶体结构。SARS-CoV-2 RBD的总体ACE2结合模式几乎与SARS-CoV RBD的相同,后者也利用ACE2作为细胞受体。
2)结构分析确定了SARS-CoV-2 RBD中对ACE2结合至关重要的残基,其中大多数是高度保守的或者与SARS-CoV RBD中的残基具有相似的侧链特性。
3)这种结构和序列上的相似性表明了SARS-CoV-2和SARS-CoV RBD之间的趋同进化,从而增强了与ACE2的结合,尽管SARS-CoV-2不会与SARS和与SARS相关的冠状病毒在进化树上高度近似。
4)研究人员还使用SARS-CoV-2 RBD分析了靶向RBD的两种SARS-CoV抗体表位,从而为深入鉴定交叉反应性抗体提供了见解。
参考文献:
Jun Lan, et al. Structure of the SARS-CoV-2 spike receptor-binding domain bound to the ACE2 receptor. Nature, 2020.
DOI:10.1038/s41586-020-2180-5
https://www.nature.com/articles/s41586-020-2180-5


















