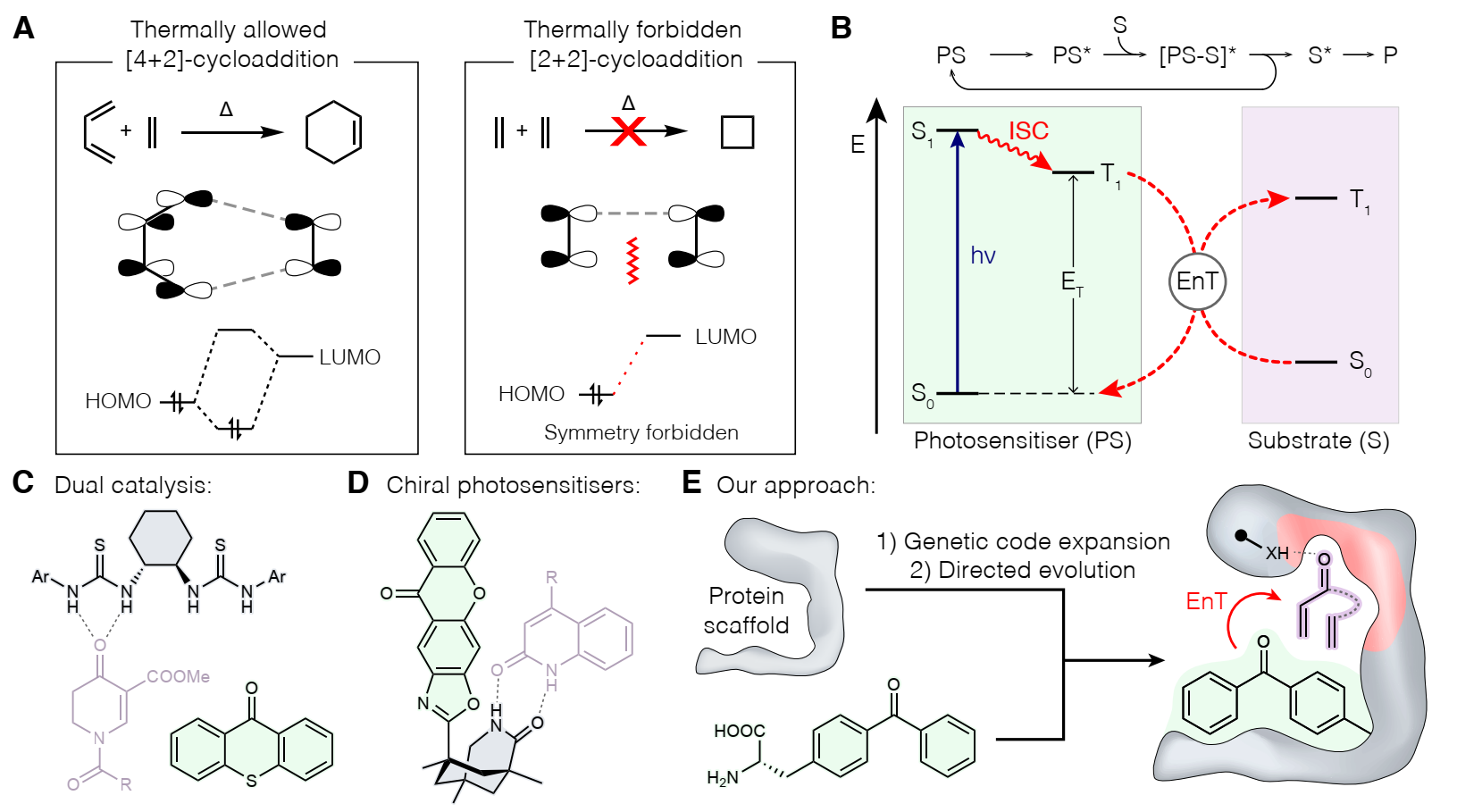
通过向蛋白中编辑新型催化剂的方式能够发展超越自然酶催化的功能,为了实现这个目标,基因编码扩展方法具有较高的前景,基因编码扩展能够向蛋白中以位点选择性的方式引入非常见的氨基酸侧链作为新型功能结构。
有鉴于此,曼彻斯特大学Anthony P. Green等报道通过扩展基因编辑方法,将酶催化领域未曾有但是有机合成领域常用的三重态能量转移催化引入酶催化领域,发展了新型光-酶催化剂,能够在温和好氧条件实现高效率的立体选择性[2+2]环加成。
本文要点:
参考文献
Trimble, J.S., Crawshaw, R., Hardy, F.J. et al. A Designed Photoenzyme for Enantioselective [2+2]-Cycloadditions. Nature (2022)
DOI: 10.1038/s41586-022-05335-3
https://www.nature.com/articles/s41586-022-05335-3