
随着遗传密码的扩展从L-α-氨基酸扩展到主链修饰和新的聚合化学,确定核糖体可以容纳哪些底物仍然是一个较大挑战。大肠杆菌核糖体在体外耐受非L-α-氨基酸,但迄今为止,很少有结构研究可以解释其具体作用,有效键形成的边界条件也仍未被揭示。鉴于此,来自美国Schrödinger公司的Ara M. Abramyan等人通过高分辨率低温电子显微镜确定了含有α-氨基酸单体的大肠杆菌核糖体的结构。
文章要点:
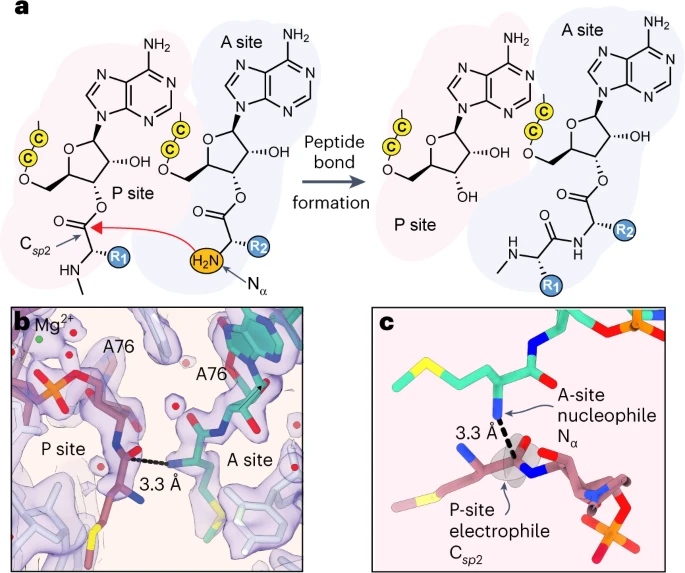
1) 该研究发现还使用元动力学模拟来定义能量表面最小值并深入了解掺入效率,发现跨不同结构类别的反应性单体有利于氨基酰基tRNA亲核试剂的构象空间(<4 Å,从距Bürgi–Dunitz 76–115°角度的肽基tRNA羰基);
2) 此外,研究还发现,落在该构象空间之外的具有自由能最小值的单体不能有效地发生反应,这一发现有望加速体内和体外核糖体合成序列定义的非肽异寡聚物。
参考资料:
Watson, Z.L., Knudson, I.J., Ward, F.R. et al. Atomistic simulations of the Escherichia coli ribosome provide selection criteria for translationally active substrates. Nat. Chem. (2023).
10.1038/s41557-023-01226-w
https://doi.org/10.1038/s41557-023-01226-w


















